利用机器学习在基因组层面预测食源性沙门氏菌的耐药性
发布时间:2024-09-14 浏览次数:964
AMR是一种全球性的健康危机,给食品生产和安全、医疗保健、公共卫生和国家经济都带来了多方面的挑战。尽管AMR本质上是一种自然进化的过程,但是抗生素的使用是耐药菌的出现和传播的一大驱动力。另外,农业环境中抗生素的滥用或者误用是造成沙门氏菌耐药的主要驱动力。随着测序技术的进步,全基因组测序(WGS)已成为常规流行病学监测的一部分。在数据分析方面,机器学习已成为分析测序平台产生的大型数据集的首选方法。然而,当前却鲜有研究将基于机器学习的数据分析技术应用在食源性致病菌的耐药性预测上。基于此,Edmund O. Benefo等人开发一种基于基因组的机器学习模型,用于预测来自鸡肉的沙门氏菌的AMR,重点关注了八种抗菌剂,包括阿莫西林-克拉维酸、氨苄西林、头孢菌素、头孢曲松、磺胺异恶唑、链霉素、四环素和头孢西丁。
研究共收集了鸡源沙门氏菌405株,取205株菌的表型和基因组信息用于机器学习模型的搭建,剩下200株作为机器模型的性能表征。研究训练了四种机器学习算法:集成分类器、随机森林、支持向量机和极端梯度提升去分析菌株中的基因存在或缺失,并将其与表型关联。在模型构建过程中,将菌株进一步按照7:3规划训练集和验证集,利用灵敏度、特异性、数据平衡准确性及科恩的κ统计值,针对每一抗生素筛选出表现性能最好的模型(表1)。然后,作者分别利用这些构建好的模型对剩余200株菌株的表型进行预测,并将其结果与ResFinder数据库中的预测结果进行比较。结果显示,模型预测的耐药性流行率从1%(头孢曲松)到65.5%(链霉素)不等。与ResFinder的预测相比,本研究的预测准确度超过95%,见图1和表2。另外,作者还分别列举出了针对每种抗生素进行预测AMR的耐药决定因子,尽管有些决定因子现今还未得到验证。
总的来说,文章证明了机器学习在预测食源性病原菌AMR的潜力,突出了机器学习方法在理解AMR机制和预测耐药模式方面的适用性和可靠性。然而,文章也有几点问题值得指出:(1)文章最后更多地关注于和ResFinder的比较,而实际上应该给出此预测方法与标准方法的比较结果;(2)研究列举了机器算法给出的权重位于前10的耐药决定因子,但是有些因子是否真的和耐药表型相关,在研究中并未得到证明。因而,未来在基于机器学习算法的耐药性预测方面,开展一些基因表达或者敲除实验去验证这些新的耐药决定因子是非常必要的。
表1 针对8种抗生素的机器学习最佳模型及其预测性能展示
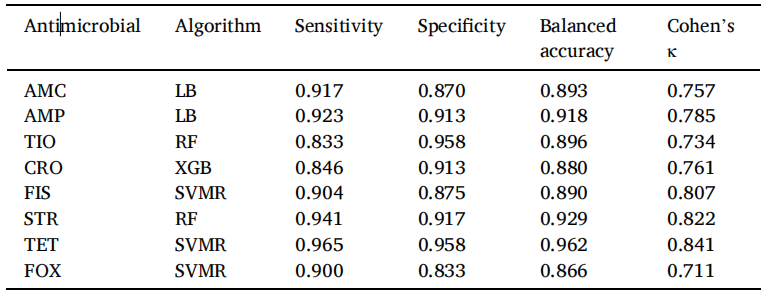
表2 基于本研究机器学习模型和基于ResFinder数据库的预测性能比较
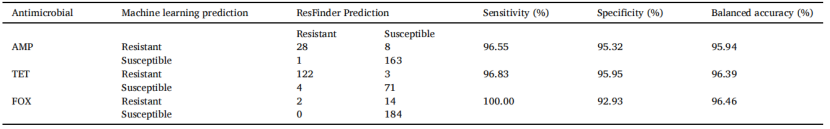
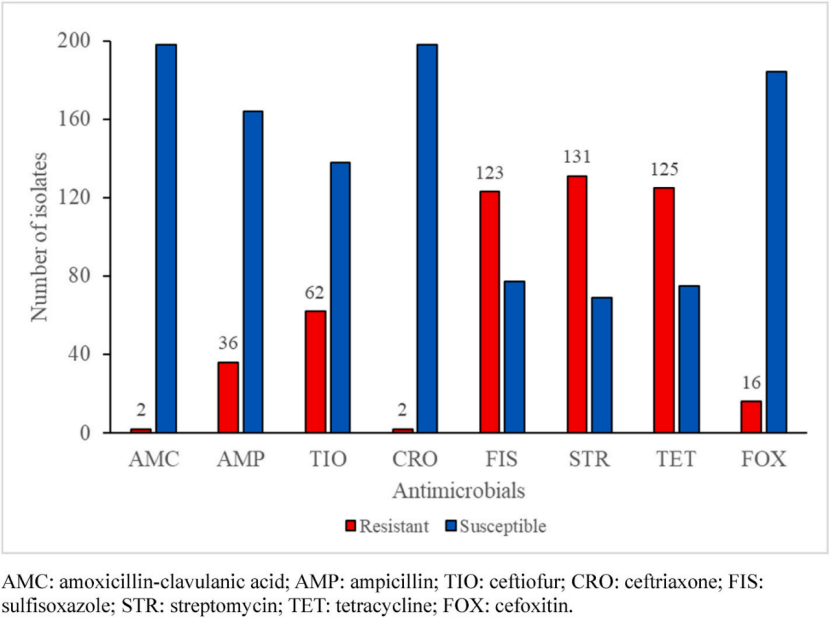
图1 所建立的机器学习模型给出的200株沙门氏菌中AMR流行率统计结果
原文链接:https://doi.org/10.1016/j.lwt.2024.116122
来源:微生物安全与健康网,作者~邹晶晶。






